Мы приняли вашу заявку, свяжемся с вами в ближайшее время.

История
Часть товаров из корзины будет перемещена в статус отложенных и не сможет быть оформлена для заказа, если вы продолжите работу в данном регионе
История

История
История
|
GeneMind, Китай
Система для высокопроизводительного секвенирования SURFSeq 5000 |
||||||||||||||||||||||||||||||||
|
SQ00023
|
|
|
|
По запросу По запросу По запросу |
|
|
|
|||||||||||||||||||||||||
|
SURFSeq 5000 — первый полногеномный NGS-секвенатор в линейке приборов, выпускаемых китайской компанией GeneMind. Благодаря наличию двух проточных ячеек и увеличенной общей производительности в 1200 Гб, на этом приборе можно отсеквенировать до 8 полных геномов человека за запуск. Также прибор рекомендуется для секвенирования экзомов и транскриптомов (около 100 за запуск) и секвенирования единичных клеток. Секвенатор работает по технологии SURF-seqTM (секвенирование осуществляется с использованием четырех меченых разными флуорофорами нуклеотидов, 3′-конец которых заблокирован (обратимые терминаторы). Генерация кластеров ДНК, секвенирование синтезом и детекция флуоресценции происходят в одном приборе, без выполнения ручных операций. В данной модели секвенатора реализована функция параллельного запуска ячеек, т.е. можно запустить секвенирование образцов на второй ячейке, во время работы прибора, не дожидаясь окончания секвенирования первой ячейки. Для удобства управления системой имеется встроенный сенсорный экран. Секвенатор также имеет полный набор программного обеспечения (с возможностью обновления) для управления системой и первичной обработки данных и встроенный аппаратный модуль для ускорения вычислений. Основные применения системы секвенирования SURFSeq 5000
Характеристики системы секвенирования SURFSeq 5000
Комплектация системы секвенирования SURFSeq 5000
|
||||||||||||||||||||||||||||||||
|
Cygnus Biosciences
Система для высокопроизводительного секвенирования P1000 |
||||||||||||||||||||||||||||||||
|
113-001-101
|
|
|
New
|
|
По запросу По запросу По запросу |
|
|
|
||||||||||||||||||||||||
|
Система для секвенирования P1000 — высокопроизводительный секвенатор от Cygnus Biosciences с производительностью до 2 ТБ данных в сутки. Благодаря технологии LumoSeq, которая является инновационным методом связывания флуоресцентной метки, обеспечивает получение длинных ридов без «молекулярных шрамов» после отщепления нуклеотидов, что повышает точность данных. При помощи технологии AttoSeal Microfluidics реализует секвенирование в ультрамалых объемах, где реагенты доставляются в ячейку через инертную масляную фазу, что снижает их расход и повышает рентабельность. В сочетании с высокоточная полимеразой и интегрированной химической реакции, при которой синтез и считывание происходят в одном процессе, улучшается детекция сигналов и обеспечивается высокая скорость работы, что гарантирует высочайшую надежность результатов. Основные применения системы секвенирования P1000:
Характеристики системы секвенирования P1000
Комплектация системы секвенирования P1000:
|
||||||||||||||||||||||||||||||||
|
GeneMind, Китай
Система для высокопроизводительного секвенирования FASTASeq 300 |
||||||||||||||||||||||||||||||||
|
SQ00020
|
|
|
|
По запросу По запросу По запросу |
|
|
|
|||||||||||||||||||||||||
|
Настольный NGS-секвенатор средней производительности для различных применений. Рекомендуется для секвенирования небольших геномов и таргетного секвенирования ампликонов и панелей генов. Имеет возможность проведения генерации кластеров ДНК и секвенирования в одном приборе, без выполнения ручных операций. FASTASeq 300 является одним из самых быстрых секвенаторов, минимальное время секвенирования и обработки данных составляет 4,5 часа. Для удобства управления системой имеется встроенный сенсорный экран. Секвенатор также имеет полный набор программного обеспечения (с возможностью обновления) для управления системой и первичной обработки данных и встроенный аппаратный модуль для ускорения вычислений. Основные применения системы для секвенирования FASTASeq 300
Характеристики системы для секвенирования FASTASeq 300
Комплектация системы для секвенирования FASTASeq 300
|
||||||||||||||||||||||||||||||||
|
Cygnus Biosciences
Система для высокопроизводительного секвенирования S100 |
||||||||||||||||||||||||||||||||
|
112-001-301
|
|
|
|
По запросу По запросу По запросу |
|
|
|
|||||||||||||||||||||||||
|
S100 — это настольный секвенатор, отличающийся компактным размером, интуитивно понятным управлением, высокой точностью секвенирования, гибкостью адаптации к различным сценариям и широким спектром выполняемых задач. Единственный прибор для массового параллельного секвенирования, в котором внедрен алгоритм коррекции ошибок (Error Correction Code), что позволяет повысить точность секвенирования в 10 раз. Генерации кластеров ДНК и секвенирование осуществляются в одном приборе, без выполнения ручных операций. Два режима секвенирования: сверхбыстрый (BitSeq), подходящий для определения количественной структуры цепи (делеции, дупликации, небольшие CNV) и сверхточный (ECC), подходящий и для детекции низкочастотных вариантов. Для удобства управления системой имеется встроенный сенсорный экран. Секвенатор также имеет полный набор программного обеспечения (с возможностью обновления) для управления системой и первичной обработки данных и встроенный аппаратный модуль для ускорения вычислений. Основные применения секвенатора S100
Характеристики секвенатора S100
Комплектация:
|
||||||||||||||||||||||||||||||||
|
GeneMind, Китай
Система для высокопроизводительного секвенирования SurfSeq Q |
||||||||||||||||||||||||||||||||
|
SQ00063
|
|
|
New
|
|
По запросу По запросу По запросу |
|
|
|
||||||||||||||||||||||||
|
SurfSeq Q — это сверхвысокопроизводительный секвенатор, новый в линейке приборов, выпускаемых компанией GeneMind. Обеспечивает беспрецедентный объём данных — 9 терабайт в день. Благодаря интеграции высокоскоростной химии, новой структурированной проточной ячейки и запатентованного алгоритма распознавания оснований на базе искусственного интеллекта, он достигает исключительной точности — ≥90% оснований при оценке Q40. Это идеальный инструмент для масштабных исследовательских проектов, таких как национальные биобанки и передовые клеточные исследования, к примеру на этом приборе можно отсеквенировать до 96 полных геномов. Основные применения системы секвенирования SurfSeq Q
Характеристики системы секвенирования SurfSeq Q
Комплектация системы секвенирования SurfSeq Q
|
||||||||||||||||||||||||||||||||
|
Oxford Nanopore Technologies
Набор для секвенирования ДНК с лигированием, Ligation Sequencing Kit |
||||||||||||||||||||||||||||||||
|
SQK-LSK109
|
|
|
хранение -20°C, транспортировка +2...+8 °C
|
|
По запросу По запросу По запросу |
|
|
|
||||||||||||||||||||||||
|
Набор предназначен для секвенирования двухцепочечной ДНК. Это наиболее универсальный набор из всех, предлагаемых Oxford Nanopore Technologies. С помощью набора Ligation Sequencing Kit можно:
Для данного набора предлагается множество протоколов пробоподготовки библиотек из различного исходного материала (геномная ДНК, плазмидная ДНК, ампликоны) и для различных задач (геномное секвенирование de novo, ресеквенирование, таргетное секвенирование, мультиплексное секвенирование и т.д.). Процесс пробоподготовки заключается в достройке концов ДНК, пришивке 3’-концевой dA и лигировании к ней адаптеров. Особенности набора
* Полные протоколы доступны на сайте www.nanoporetech.com для зарегистрированных пользователей-участников Nanopore Community. Набор оптимизирован для высокопроизводительного секвенирования. Для максимальной производительности рекомендуется получать библиотеку из 100-200 фмоль высокомолекулярной ДНК. Также важна степень чистоты ДНК, загрязнения существенно снижают производительность. Рекомендуется использовать наноспектрофотометр для оценки степени чистоты ДНК по следующим критериям:
Можно секвенировать библиотеки, полученные с помощью Ligation Sequencing Kit из меньшего количества ДНК или ДНК низкого качества, но это существенно уменьшает производительность секвенирования. Частично можно улучшить библиотеку с помощью стадии ПЦР: происходит обогащение библиотеки при малом стартовом количестве ДНК, а также снижается влияние примесей за счёт разведения образца. Но, если ПЦР является неотъемлемой частью пробоподготовки, рекомендуется использовать PCR Sequencing Kit или Rapid PCR Barcoding Kit. Аналогично, если планируется регулярно секвенировать ампликоны или кДНК, рекомендуется использовать соответствующие наборы — PCR Sequencing Kit или, для секвенирования кДНК, cDNA-PCR Sequencing Kit и Direct cDNA Sequencing Kit. Набор Ligation Sequencing Kit может быть использован для мультиплексного секвенирования. Для этого необходимы дополнительные наборы Native Barcoding Expansion 1-12 и 13-24 (для мультиплексирования без стадии ПЦР) или PCR Barcoding Expansion 1-12 и 1-96 (для мультиплексирования со стадией ПЦР). Дополнительные расходные материалы и реактивы:
Опционально:
Дополнительное оборудование:
Опционально:
|
||||||||||||||||||||||||||||||||
|
A43540исг
|
Стандартная цена: 8 091,= USD.
Скидка 50%.
Стандартная цена: 685 158,=.
Скидка 50%.
Стандартная цена: .
Скидка 50%.
|
|
|
|
|
|
||||||||||||||||||||||||||
|
Набор чипов содержит 4 штрихкодированных чипа для секвенирования с помощью систем Ion S5 и Ion S5 XL. Чип Ion 540 Chip обнаруживает встраивание оснований под действием полимеразы без использования флуоресценции, благодаря чему позволяет проводить секвенирование 60-80 млн фрагментов длиной 200 п.н. в в течение всего 2,5 часов. Поддерживает различные исследовательские приложения, включая секвенирование до трех экзомов человека, профилирование транскриптома и целевое повторное секвенирование генных панелей. |
||||||||||||||||||||||||||||||||
|
GeneMind, Китай
Система для высокопроизводительного секвенирования FASTASeq S |
||||||||||||||||||||||||||||||||
|
SQ00075
|
|
|
New
|
|
По запросу По запросу По запросу |
|
|
|
||||||||||||||||||||||||
|
FASTASeq S — сверхвысокопроизводительная платформа секвенирования. Время выполнения всего процесса — от подготовки финальной библиотеки до вывода финальных данных — составляет всего 2 часа (SE50), 3 часа (SE100) и 6-8 часа (PE150). Эта производительность достигается благодаря синергетической интеграции основных технологий:
В основе работы секвенатора лежит технология SURF-seq, использующая секвенирование синтезом с обратимыми терминаторами (четыре нуклеотида с разными флуорофорами). Процессы генерации ДНК-кластеров, секвенирования и детекции флуоресценции полностью автоматизированы и интегрированы в один прибор. Основные применения системы секвенирования FASTASeq S
Характеристики системы секвенирования FASTASeq S
Комплектация системы секвенирования FASTASeq S
|
||||||||||||||||||||||||||||||||
|
Oxford Nanopore Technologies
Набор для прямого секвенирования РНК, Direct RNA Sequencing Kit |
||||||||||||||||||||||||||||||||
|
SQK-RNA002
|
|
|
хранение -20°C, транспортировка +2...+8 °C
|
|
По запросу По запросу По запросу |
|
|
|
||||||||||||||||||||||||
|
Набор предназначен для прямого секвенирования РНК без стадии обратной транскрипции. С помощью набора Direct RNA Sequencing Kit можно:
Особенности набора
* Полные протоколы доступны на сайте www.nanoporetech.com для зарегистрированных пользователей-участников Nanopore Community.
Набор предназначен для работы с молекулами РНК, обладающими 3’-концевыми полиА –структурами — эукариотическими мРНК, полиаденилированными вирусными РНК или РНК, обработанными наборами для полиаденилирования. Для РНК без полиА-концов можно синтезировать пользовательские адаптеры, комплементарные 3’-концам (например, для тРНК или 16S-РНК). В отличие от протоколов секвенирования кДНК, в случае использования набора Direct RNA Sequencing Kit происходит секвенирование нативных молекул РНК; только они проходят через поры проточной ячейки. Стадия обратной транскрипции при пробоподготовке опциональна, но настоятельно рекомендуется — это повышает производительность секвенирования. Набор Direct RNA Sequencing Kit незаменим в случае анализа модифицированных нуклеотидов. Если задача выявления таких нуклеотидов не ставится, рекомендуются наборы для секвенирования кДНК, так как они обеспечивают более высокую производительность.
Дополнительные расходные материалы и реактивы:
Дополнительное оборудование:
Опционально:
|
||||||||||||||||||||||||||||||||
|
001.24
|
||||||||||||||||||||||||||||||||
|
Набор для приготовления библиотек для секвенирования определённой (+ или -) цепи мРНК, включающий все необходимые компоненты.
Эти библиотеки предназначены для следующих задач:
В состав набора входят все необходимые компоненты для подготовки библиотеки:
Входящий в набор Poly(A) RNA Selection Kit (его можно также приобрести отдельно) обеспечивает эффективную элиминацию рибосомальной РНК. Доля ридов рРНК составляет не более 0.001%. |
||||||||||||||||||||||||||||||||
|
009.08
|
||||||||||||||||||||||||||||||||
|
Продукт доступен до 31.12.2019.
Набор для приготовления цепь-специфичных библиотек для секвенирования РНК, включающий все необходимые компоненты. Библиотеки предназначены для следующих задач:
С помощью набора SENSE можно получать библиотеки для секвенирования только на платформе Illumina. Исходным материалом для получения библиотек является РНК после удаления рибосомальной РНК или обогащения поли-А+. Для удаления рибосомальной РНК из образца можно использовать RiboCop rRNA Depletion Kit. Для работы с набором предлагаются два протокола - для интактной РНК и деградированной РНК (например, из FFPЕ-образцов). В состав набора входят все необходимые компоненты для подготовки библиотеки:
|
||||||||||||||||||||||||||||||||
|
Oxford Nanopore Technologies
Набор для мультиплексного секвенирования ДНК с баркодированием образцов методом ПЦР, PCR Barcoding Kit |
||||||||||||||||||||||||||||||||
|
SQK-PBK004
|
|
|
хранение -20°C
|
|
По запросу По запросу По запросу |
|
|
|
||||||||||||||||||||||||
|
Мультиплексное секвенирование с баркодированием удобно, если количество сиквенсных данных, получаемое с одного образца, существенно меньше производительности проточной ячейки. В таком случае имеет смысл объединять образцы в одну библиотеку и секвенировать их одновременно. Это также снижает стоимость секвенирования каждого образца.
Набор PCR Barcoding Kit удобен в тех случаях, когда:
Особенности набора:
* Полные протоколы доступны на сайте
www.nanoporetech.com для зарегистрированных пользователей-участников Nanopore Community. Пробоподготовка заключается в опциональной фрагментации геномной ДНК с последующей достройкой концов, лигировании адаптеров к достроенным концам и ПЦР с праймерами, содержащими бар-коды. В состав набора входит 12 пар бар-кодированных праймеров. В конечном итоге образцы после ПЦР объединяются, и к смеси добавляются сиквенсные адаптеры. Длины фрагментов, получаемых в ходе ПЦР, зависят от длины исходной матрицы и от процессивности ДНК-полимеразы. Максимальная длина рида составляет в среднем около 2 тыс п.н. Пользователь также может использовать метод ПЦР с 2 парами праймеров (nested PCR); при этом одна пара праймеров - пользовательские праймеры к определённой последовательности ДНК, а вторая - бар-кодированные праймеры из набора. 5’-концы пользовательских праймеров должны быть комплементарны 3’-концам бар-кодированных праймеров. Последовательности бар-кодов автоматически распознаются программой для интерпретации данных секвенирования, и риды группируются в соответствии с бар-кодами. Ещё одно преимущество набора — если исходная ДНК загрязнена, то в ходе ПЦР существенно снижается концентрация примесей, которые могут ингибировать процесс получения библиотеки. Если количество исходной ДНК менее 1 нг — рекомендуется предварительная полногеномная амплификация. Дополнительные расходные материалы и реактивы:
Опционально:
Дополнительное оборудование:
|
||||||||||||||||||||||||||||||||
|
006.08
|
||||||||||||||||||||||||||||||||
|
Набор для приготовления библиотек для секвенирования определённой (+ или -) цепи мРНК, включающий все необходимые компоненты.
Эти библиотеки предназначены для следующих задач:
В состав набора входят все необходимые компоненты для подготовки библиотеки:
Входящий в набор Poly(A) RNA Selection Kit (его можно также приобрести отдельно) обеспечивает эффективную элиминацию рибосомальной РНК. Доля ридов рРНК составляет не более 0.001%. |
||||||||||||||||||||||||||||||||
|
A40271
|
||||||||||||||||||||||||||||||||
|
Набор содержит материалы, необходимые для проведения двух инициализаций и автоматического секвенирования двух многополосных чипов за запуск с помощью интегрированного секвенатора Ion Torrent Genexus. Набор автоматизирует секвенирование библиотек Ion AmpliSeq или Ion AmpliSeq HD с длиной чтений 200-400 п.н. Содержимое и условия хранения
|
||||||||||||||||||||||||||||||||
|
Oxford Nanopore Technologies
Набор для секвенирования ДНК со стадией ПЦР, PCR Seqiencing Kit |
||||||||||||||||||||||||||||||||
|
SQK-PSK004
|
|
|
хранение -20°C, транспортировка +2...+8 °C
|
|
По запросу По запросу По запросу |
|
|
|
||||||||||||||||||||||||
|
Набор предназначен для получения сиквенсной библиотеки из геномной ДНК в тех случаях, когда количество стартового материала ограничено. С помощью набора PCR Sequencing Kit можно:
Особенности набора:
* Полные протоколы доступны на сайте www.nanoporetech.com для зарегистрированных пользователей-участников Nanopore Community. Пробоподготовка заключается в опциональной фрагментации ДНК с последующей достройкой концов до тупых и присоединением 3’-dA. К 3’-dA лигируются олигонуклеотиды, содержащие сайты связывания ПЦР-праймеров. ДНК амплифицируется, и к ампликонам присоединяются сиквенсные адаптеры. Стадия ПЦР зависит от исходного количества ДНК и желаемой длины ампликонов. Рекомендуемый протокол ПЦР рассчитан на 100 нг исходной ДНК. Длина рида составляет в среднем около 2 килобаз и зависит не столько от процессивности Taq-полимеразы, сколько от особенностей пробоподготовки. Но при этом максимальная длина рида выше, чем при использовании набора Rapid PCR Barcoding Kit и может достигать 10 килобаз. Пользователь также может использовать метод ПЦР с 2 парами праймеров (nested PCR); при этом одна пара праймеров — пользовательские праймеры к определённой последовательности ДНК, а вторая — бар-кодированные праймеры из набора. 5’-концы пользовательских праймеров должны быть комплементарны 3’-концам бар-кодированных праймеров. Набор позволяет работать с ДНК, содержащей примеси, которые снижают эффективность получения библиотеки. Это происходит благодаря стадии ПЦР. Но необходимо учитывать, что амплификация разных фрагментов может идти с разной эффективностью. Если исходное количество ДНК менее 1 пг, рекомендуется полногеномная амплификация. В состав PCR Sequencing Kit также входит набор Flow Cell Priming Kit для загрузки библиотеки в поточную ячейку. Условия хранения: -20 °С. Дополнительные расходные материалы и реактивы:
Опционально:
Дополнительное оборудование:
Опционально:
|
||||||||||||||||||||||||||||||||
|
375.17.036
|
||||||||||||||||||||||||||||||||
|
AMGene Dye v3.1 — готовый набор реагентов, предназначенный для циклического секвенирования по методу Сенгера с использованием флуоресцентно-меченых терминирующих трифосфатов. Реакция секвенирования проводится путем циклической элонгации ДНК с нуклеотид-специфическим терминированием флуоресцентными аналогами нуклеотидов. Набор представляет собой более экономичную и качественную альтернативу реагентам BigDye Terminator v3.1, Thermo Fisher. Основные характеристики набора AMGene Dye v3.1
Состав набора и условия хранения
Хранение компонентов набора: от -18 до -25 °С в защищенном от света месте. Транспортирование: при температуре не выше -10 °С. Гарантийный срок годности набора: в течении 12 месяцев с даты изготовления. |
||||||||||||||||||||||||||||||||
|
Oxford Nanopore Technologies
Набор для секвенирования гена 16S-РНК, 16S Barcoding Kit |
||||||||||||||||||||||||||||||||
|
SQK-RAB204
|
|
|
хранение -20°C
|
|
По запросу По запросу По запросу |
|
|
|
||||||||||||||||||||||||
|
Набор предназначен для секвенирования бактериальных генов, кодирующих рибосомальную РНК 16S, являющуюся видоспецифичной. С помощью набора 16S Barcoding Kit можно:
Объединение нескольких образцов, или мультиплексирование, возможно благодаря баркодированию разных ДНК. Это удобно в тех случаях, когда требуемый выход данных при секвенировании одного образца намного меньше максимального выхода, получаемого с одной проточной ячейки. В состав набора входит 12 бар-кодов, таким образом, в одну библиотеку можно загрузить до 12 образцов ДНК. Это позволяет снизить стоимость секвенирования одного образца почти в 10 раз. Особенности набора:
* Полные протоколы доступны на сайте www.nanoporetech.com для зарегистрированных пользователей-участников Nanopore Community. Пробоподготовка заключается в амплификации гена 16S со специфических праймеров (27F и 1492R), содержащих бар-коды. Затем к 5’-концам ампликонов лигируются сиквенсные адаптеры. Анализ результатов секвенирования библиотеки, полученной с помощью 16S Barcoding Kit, проводится на платформе EPI2ME с помощью специального ПО, сортирующего риды в соответствии с баркодированием и идентифицирующего их видовую принадлежность. В состав 16S Barcoding Kit также входит набор Flow Cell Priming Kit для загрузки библиотеки в поточную ячейку. Условия хранения: - 20 °С. Дополнительные расходные материалы и реактивы:
Дополнительное оборудование:
Опционально:
|
||||||||||||||||||||||||||||||||
|
SQK-RPB004
|
|
|
хранение -20°C, транспортировка +2...+8 °C
|
|
По запросу По запросу По запросу |
|
|
|
||||||||||||||||||||||||
|
Набор предназначен для быстрого и простого получения сиквенсной библиотеки, включающей до 12 различных образцов ДНК, в тех случаях, когда количество ДНК
ограничено. С помощью набора Rapid PCR Barcoding Kit можно:
Особенности набора:
* Полные протоколы доступны на сайте www.nanoporetech.com для зарегистрированных пользователей-участников Nanopore Community. Набор предназначен для исследований, требующих простой пробоподготовки и быстрого результата, но не максимальной производительности секвенирования. Пробоподготовка заключается в расщеплении ДНК транспозазой и присоединении к фрагментам олигонуклеотидов, содержащих сайты связывания ПЦР-праймеров. Затем каждый образец ДНК амплифицируется с помощью баркодированных праймеров. Баркодированные ампликоны объединяются, и к ним присоединяются сиквенсные адаптеры. Длина ампликонов в среднем составляет около 2 килобаз и не зависит от длины исходной ДНК. Это связано в большей степени с особенностями пробоподготовки, а не с процессивностью Taq-полимеразы. Поэтому для пользователей, работающих с малым исходным количеством геномной ДНК и желающих получить риды максимальной длины, рекомендуется набор Ligation Sequencing Kit и протокол Low input genomic DNA with PCR. Набор позволяет работать с ДНК, содержащей примеси, которые снижают эффективность получения библиотеки. Это происходит благодаря стадии ПЦР. Но необходимо учитывать, что амплификация разных фрагментов может идти с разной эффективностью. Если исходное количество ДНК менее 1 пг, рекомендуется полногеномная амплификация. Сортировка ридов в соответствии с баркодами производится с помощью программных инструментов, доступных в рамках платформы EPI2ME. В состав Rapid PCR Barcoding Kit также входит набор Flow Cell Priming Kit для загрузки библиотеки в поточную ячейку. Условия хранения: -20 °С. Дополнительные расходные материалы и реактивы:
Дополнительное оборудование:
|
||||||||||||||||||||||||||||||||
|
Oxford Nanopore Technologies
Набор для быстрого мультиплексного секвенирования ДНК, Rapid Barcoding Kit |
||||||||||||||||||||||||||||||||
|
SQK-RBK004
|
|
|
хранение -20°C
|
|
По запросу По запросу По запросу |
|
|
|
||||||||||||||||||||||||
|
Набор предназначен для быстрого и простого получения сиквенсной библиотеки, включающей до 12 различных образцов ДНК. С помощью набора Rapid Barcoding Kit можно:
Объединение нескольких образцов, или мультиплексирование, возможно благодаря бар-кодированию разных ДНК. Это удобно в тех случаях, когда требуемый выход данных при секвенировании одного образца намного меньше максимального выхода, получаемого с одной проточной ячейки. В состав набора входит 12 бар-кодов, таким образом, в одну библиотеку можно загрузить до 12 образцов ДНК. Это позволяет снизить стоимость секвенирования одного образца почти в 10 раз.
Особенности набора:
* Полные протоколы доступны на сайте www.nanoporetech.com для зарегистрированных пользователей-участников Nanopore Community. Набор предназначен для исследований, требующих простой пробоподготовки и быстрого результата, но не максимальной производительности секвенирования. Так как количество стадий пипетирования и время подготовки минимальны, набор даёт возможность получения длинных ридов, но макcимальная длина зависит от качества исходной ДНК. Поэтому для лучшего результата рекомендуется использовать ДНК со средней длиной фрагментов не менее 30 гигабаз. Для выделения высокомолекулярной геномной ДНК можно использовать набор Blood & Cell Culture DNA Mini Kit (Qiagen). Пробоподготовка заключается в расщеплении ДНК транспозазой и присоединении к фрагментам бар-кодов. Баркодированные фрагменты ДНК объединяются, и к ним присоединяются сиквенсные адаптеры. Рекомендуемое исходное количество ДНК — не менее 400 нг. Возможно получение библиотеки из меньшего количества ДНК, а также из фрагментированной ДНК, но производительность будет ниже. Для получения библиотеки из меньших количеств ДНК рекомендуется набор PCR Sequencing Kit и протоколы Low Input и Rapid Low Input*. Так как при получении библиотеки отсутствует стадия ПЦР, данный набор пригоден для выявления модифицированных нуклеотидов. Сортировка ридов в соответствии с баркодами производится с помощью программных инструментов, доступных в рамках платформы EPI2ME. В состав Rapid Barcoding Kit также входит набор Flow Cell Priming Kit для загрузки библиотеки в поточную ячейку. Сиквенсные адаптеры RAP, входящие в набор, также могут быть приобретены в виде отдельного набора EXP-RAP001.
Условия хранения: -20 °С.
Дополнительные расходные материалы и реактивы:
Опционально:
Дополнительное оборудование:
Опционально:
|
||||||||||||||||||||||||||||||||
|
Oxford Nanopore Technologies
Набор для секвенирования кДНК со стадией ПЦР, PCR-cDNA Sequencing Kit |
||||||||||||||||||||||||||||||||
|
SQK-PCS109
|
|
|
хранение -20°C
|
|
По запросу По запросу По запросу |
|
|
|
||||||||||||||||||||||||
|
Набор для секвенирования кДНК со стадией ПЦР PCR-cDNA Sequencing Kit предназначен для секвенирования РНК через стадию синтеза кДНК с последующей ПЦР. С помощью набора PCR-cDNA Sequencing Kit можно:
В состав набора входит Pro Flow Cell Priming Kit для загрузки библиотек в ячейку, совместимый с ячейками для PromethION Особенности набора:
Набор рассчитан на работу с минимальным количеством поли-А+ РНК, для получения библиотеки достаточно всего 1 нг. В случае невозможности выделения полиА+-фракции допустимо использование суммарной РНК (рекомендуется не менее 50 нг), но в этом случае требуется оптимизация пробоподготовки. Пробоподготовка заключается в синтезе комплементарной цепи кДНК с помощью обратной транскрипции. Обогащение библиотеки полноразмерными копиями происходит за счёт переключения цепей при обратной транскрипции (strand-switching). Затем происходит синтез двухцепочечной кДНК с помощью ПЦР, в реакции используются специфические праймеры для последующего безлигазного присоединения сиквенсных адаптеров. При работе с PCR-cDNA Sequencing Kit предусмотрена возможность мультиплексирования с помощью набора PCR Barcoding Expansion (SQK-PBK004). В одну библиотеку может быть загружено до 12 образцов кДНК. Дополнительные расходные материалы и реактивы:
Дополнительное оборудование:
Опционально:
|
||||||||||||||||||||||||||||||||
|
Oxford Nanopore Technologies
Набор для прямого секвенирования кДНК, Direct cDNA Sequencing Kit |
||||||||||||||||||||||||||||||||
|
SQK-DCS109
|
|
|
хранение -20°C
|
|
РУ
Регистрационное удостоверение на медицинское изделие
Росздравнадзора
|
По запросу По запросу По запросу |
|
|
|
|||||||||||||||||||||||
|
Direct cDNA Sequencing Kit — набор для прямого секвенирования кДНК. Набор предназначен для секвенирования РНК через стадию синтеза кДНК без стадии ПЦР. С помощью набора Direct DNA Sequencing Kit можно:
Особенности набора:
Набор рассчитан на работу с поли-А+ РНК, но возможно также использование РНК со специфическими 3’-концевыми последовательностями (например, вирусной РНК). Для этого необходимы олигонуклеотиды, комплементарные этим последовательностям. Пробоподготовка заключается в синтезе комплементарной цепи кДНК с помощью обратной транскрипции. Обогащение библиотеки полноразмерными копиями происходит за счёт переключения цепей при обратной транскрипции (strand-switching). Затем цепь РНК расщепляется, и достраивается вторая цепь кДНК. Сиквенсные адаптеры лигируются на двухцепочечную кДНК (в нанопоры может проходить любая из цепей кДНК). При работе с Direct cDNA Sequencing Kit предусмотрена возможность мультиплексирования с помощью наборов Native Barcoding Expansion 1-12 (EXP-NBD104) или Native Barcoding Expansion 13-24 (EXP-NBD105). В одну библиотеку может быть загружено до 12 образцов кДНК. Дополнительные расходные материалы и реактивы:
Дополнительное оборудование:
Опционально:
|
||||||||||||||||||||||||||||||||
|
4336697
|
||||||||||||||||||||||||||||||||
|
5-кратный буфер для секвенирования по Сэнгеру. Оптимизирован для использования с наборами для секвенирования BigDye Terminator v1.1 и v3.1.
|
||||||||||||||||||||||||||||||||
|
375.17.043
|
|
1000 мкл
|
хранение -20°C, транспортировка -20°C
|
|
По запросу По запросу По запросу |
|
|
|
||||||||||||||||||||||||
|
Пятикратный (5Х) буфер для оптимизации реакции циклического секвенирования по Сенгеру, предназначен для использования с наборами для циклического секвенирования по Сенгеру AMGene Dye v3.1, BigDye Terminator v1.1 и v3.1. |
||||||||||||||||||||||||||||||||
|
012.24A
|
|
24 образца
|
|
По запросу По запросу По запросу |
|
|
|
|||||||||||||||||||||||||
|
Набор предназначен для получения библиотек, содержащих 3’-концевые последовательности полиА+мРНК, для секвенирования на платформе Ion Torrent. При этом риды генерируются в направлении 3’-концов, отражая истинную последовательность мРНК.
Для получения библиотеки достаточно 5 нг суммарной РНК, в том числе деградированной (например, из FFPE-образцов). Специфичность цепи при секвенировании составляет не менее 99.9%, что позволяет картировать риды на соответствующую цепь геномной ДНК. Это позволяет выявлять антисмысловые транскрипты и перекрывающиеся гены. Процесс получения библиотеки занимает около 4.5 часов. Поскольку каждый транскрипт даёт только один фрагмент, не требуется нормализация по длине. Это позволяет количественно анализировать экспрессию генов с высокой точностью. Для получения мультиплексных библиотек в состав наборов входит 24 баркода (набор А или набор В). Таким образом, в одну библиотеку можно загрузить до 48 образцов. |
||||||||||||||||||||||||||||||||
|
012.24B
|
|
24 образца
|
|
По запросу По запросу По запросу |
|
|
|
|||||||||||||||||||||||||
|
Набор предназначен для получения библиотек, содержащих 3’-концевые последовательности полиА+мРНК, для секвенирования на платформе Ion Torrent. При этом риды генерируются в направлении 3’-концов, отражая истинную последовательность мРНК.
Для получения библиотеки достаточно 5 нг суммарной РНК, в том числе деградированной (например, из FFPE-образцов). Специфичность цепи при секвенировании составляет не менее 99.9%, что позволяет картировать риды на соответствующую цепь геномной ДНК. Это позволяет выявлять антисмысловые транскрипты и перекрывающиеся гены. Процесс получения библиотеки занимает около 4.5 часов. Поскольку каждый транскрипт даёт только один фрагмент, не требуется нормализация по длине. Это позволяет количественно анализировать экспрессию генов с высокой точностью. Для получения мультиплексных библиотек в состав наборов входит 24 баркода (набор А или набор В). Таким образом, в одну библиотеку можно загрузить до 48 образцов. |
||||||||||||||||||||||||||||||||
|
Oxford Nanopore Technologies
Набор для быстрого секвенирования геномной ДНК, Rapid Sequencing Kit |
||||||||||||||||||||||||||||||||
|
SQK-RAD004
|
|
|
хранение -20°C
|
|
По запросу По запросу По запросу |
|
|
|
||||||||||||||||||||||||
|
Набор предназначен для быстрого получения библиотеки из геномной ДНК с помощью простого 2-шагового протокола, пробоподготовка занимает не более 10 минут. Процесс основан на транспозазном расщеплении ДНК с последующей пришивкой сиквенсных адаптеров.
Особенности набора:
Набор предназначен для исследований, требующих простой пробоподготовки и быстрого результата, но не максимальной производительности секвенирования. Так как количество стадий пипетирования и время подготовки минимальны, набор даёт возможность получения длинных ридов, но макcимальная длина зависит от качества исходной ДНК. Поэтому для лучшего результата рекомендуется использовать ДНК со средней длиной фрагментов не менее 30 гигабаз. Для выделения высокомолекулярной геномной ДНК можно использовать набор Blood & Cell Culture DNA Mini Kit (Qiagen). Рекомендуемое исходное количество ДНК — не менее 400 нг. Возможно получение библиотеки из меньшего количества ДНК, а также из фрагментированной ДНК, но производительность будет ниже. Для получения библиотеки из меньших количеств ДНК рекомендуется набор PCR Sequencing Kit и протоколы Low Input и Rapid Low Input. Так как при получении библиотеки отсутствует стадия ПЦР, данный набор пригоден для выявления модифицированных нуклеотидов. Дополнительные расходные материалы:
Дополнительное оборудование:
Опционально:
|
||||||||||||||||||||||||||||||||
|
4337450
|
||||||||||||||||||||||||||||||||
|
Набор реагентов для секвенирования по Сэнгеру. Оптимизирован для более длинных прочтений (свыше 500 п.н.).
|
||||||||||||||||||||||||||||||||
|
4404312
|
|
1 набор
|
хранение -25...-15°C, транспортировка сухой лед
|
|
122 156, руб.
122 156, руб. RUB
|
|
|
|
||||||||||||||||||||||||
|
Набор стандартов секвенирования v. 3.1, содержит в себе стандартную последовательность из 1200 пар нуклеотидов. Предназначен для проведения спектральной калибровки и проверочного запуска на генетических анализаторах 3500, 3500xl и SeqStudio. Упаковка содержит 4 пробирки с лиофилизированным стандартом.
|
||||||||||||||||||||||||||||||||
|
007.08A
|
||||||||||||||||||||||||||||||||
|
Наборы SENSE mRNA-Seq Barcode Kit for Ion Torrent предназначены для мультиплексирования при получении библиотек с помощью наборов SENSE для РНК-секвенирования на платформе Ion Torrent.
В каждый набор входит 12 баркодов, позволяющих объединять в библиотеку до 8 или 24 образцов. Пришивка баркодов происходит на стадии обратной транскрипции-лигирования. Баркоды представляют собой 17-нуклеотидные последовательности на 5’-концах ридов; 9 уникальных нуклеотидов фланкированы справа и слева константными 4-нуклеотидными структурами (TCAG-баркод-CGAT). |
||||||||||||||||||||||||||||||||
|
007.08В
|
||||||||||||||||||||||||||||||||
|
Наборы SENSE mRNA-Seq Barcode Kit for Ion Torrent предназначены для мультиплексирования при получении библиотек с помощью наборов SENSE для РНК-секвенирования на платформе Ion Torrent.
В каждый набор входит 12 баркодов, позволяющих объединять в библиотеку до 8 или 24 образцов. Пришивка баркодов происходит на стадии обратной транскрипции-лигирования. Баркоды представляют собой 17-нуклеотидные последовательности на 5’-концах ридов; 9 уникальных нуклеотидов фланкированы справа и слева константными 4-нуклеотидными структурами (TCAG-баркод-CGAT). |
||||||||||||||||||||||||||||||||
|
091.24
|
|
24 запуска
|
|
По запросу По запросу По запросу |
|
|
|
|||||||||||||||||||||||||
|
090.24
|
|
24 запуска
|
|
По запросу По запросу По запросу |
|
|
|
|||||||||||||||||||||||||
|
Oxford Nanopore Technologies
Набор для секвенирования ДНК в полевых условиях, Field Sequencing Kit |
||||||||||||||||||||||||||||||||
|
SQK-LRK001
|
|
|
|
По запросу По запросу По запросу |
|
|
|
|||||||||||||||||||||||||
|
Набор предназначен для получения библиотеки из геномной ДНК в полевых условиях, когда ограничена возможность хранения реагентов при низких температурах. Процесс пробоподготовки основан на транспозазном расщеплении ДНК с последующей пришивкой сиквенсных адаптеров и занимает не более 10 минут.
Особенности набора:
Набор оптимизирован для хранения при температуре окружающей среды, так как реагенты в его составе находятся в лиофилизированном виде. Количество стадий пипетирования и время подготовки минимальны, поэтому набор даёт возможность получения длинных ридов, но максимальная длина зависит от качества исходной ДНК. Рекомендуемое исходное количество ДНК — не менее 400 нг. Возможно получение библиотеки из меньшего количества ДНК, а также из фрагментированной ДНК, но производительность будет ниже. Так как при получении библиотеки отсутствует стадия ПЦР, данный набор пригоден для выявления модифицированных нуклеотидов. В состав Field Sequencing Kit также входит набор Flow Cell Priming Kit для загрузки библиотеки в поточную ячейку. Дополнительные расходные материалы:
|
||||||||||||||||||||||||||||||||
|
093.03
|
||||||||||||||||||||||||||||||||
|
063.02
|
||||||||||||||||||||||||||||||||
|
094.03
|
||||||||||||||||||||||||||||||||
|
4336799
|
|
1 набор
|
|
По запросу По запросу По запросу |
|
|
|
|||||||||||||||||||||||||
|
A27760
|
|
|
|
По запросу По запросу По запросу |
|
|
|
|||||||||||||||||||||||||
|
|
||||||||||||||||||||||||||||||||
|
A27766
|
|
|
|
По запросу По запросу По запросу |
|
|
|
|||||||||||||||||||||||||
|
|
||||||||||||||||||||||||||||||||
|
A27762
|
|
|
хранение комнатная температура
|
|
По запросу По запросу По запросу |
|
|
|
||||||||||||||||||||||||
|
|
||||||||||||||||||||||||||||||||
|
ГМ00010
|
|
|
New
|
|
РУ
Регистрационное удостоверение на медицинское изделие
Росздравнадзора
|
По запросу По запросу По запросу |
|
|
|
|||||||||||||||||||||||
|
Прибор зарегистрирован в РФ в качестве медицинского изделия. РЗН 2025/24616. В комплект поставки входит:
Настольный высокопроизводительный NGS-секвенатор для диагностических целей. Имеет возможность проведения генерации кластеров ДНК и секвенирования в одном приборе, без выполнения ручных операций. Для удобства управления системой имеется встроенный сенсорный экран. Секвенатор также имеет полный набор программного обеспечения (с возможностью обновления) для управления системой и первичной обработки данных и встроенный аппаратный модуль для ускорения вычислений. Основные области применения NGS-секвенатора Геноскан 4000
Характеристики NGS-секвенатора Геноскан 4000
Комплектация секвенатора Геноскан 4000
|
||||||||||||||||||||||||||||||||
|
SQ00003
|
|
|
|
По запросу По запросу По запросу |
|
|
|
|||||||||||||||||||||||||
|
Настольный высокопроизводительный NGS-секвенатор для различных применений. Имеет возможность проведения генерации кластеров ДНК и секвенирования в одном приборе, без выполнения ручных операций. Для удобства управления системой имеется встроенный сенсорный экран. Секвенатор также имеет полный набор программного обеспечения (с возможностью обновления) для управления системой и первичной обработки данных и встроенный аппаратный модуль для ускорения вычислений Основные применения высокопроизводительного NGS-секвенатора GenoLab M
Характеристики NGS-секвенатора GenoLab M
Комплектация секвенатора GenoLab M
|
||||||||||||||||||||||||||||||||
|
SY-410-1003
|
|
|
|
РУ
Регистрационное удостоверение на медицинское изделие
Росздравнадзора
|
По запросу По запросу По запросу |
|
|
|
||||||||||||||||||||||||
|
Товар временно недоступен. Аналоги по запросу ниже. Настольный интегрированный инструмент для NGS, включающий в одном приборе блоки для подготовки образцов (образования кластеров), секвенирования и обработки данных и интерфейс для управления. Принцип секвенирования основан на технологии Solexa, включающей обогащение методом bridge-ПЦР на проточном чипе, секвенирование методом синтеза (SBS) с использованием флуоресцентно-меченных нуклеотидов и детекцию света флуоресценции от кластеров ДНК. Основные применения:
|
||||||||||||||||||||||||||||||||
|
PY-NSR001
|
||||||||||||||||||||||||||||||||
|
Аналоги:
Аналоги
|
||||||||||||||||||||||||||||||||
|
PolyseqOne — это портативная система нанопорового секвенирования средней производительности, позволяющая проводить анализ в режиме реального времени. Компания Polyseq Biotechnology разработала собственные нанопоровые белки, наборы для подготовки библиотек и секвенирования, конструкцию чипа, программную инженерию и алгоритмы глубокого обучения. Нанопоровые белки закреплены на полимерной мембране, которая окружена электролитным раствором с обеих сторон. Ионы электролита проходят через нанопору, создавая постоянный электрический ток. Молекулы ДНК или РНК обладают электрическим зарядом, и под действием электрического поля одноцепочечные молекулы проходят через нанопору от отрицательно заряженной стороны (cis) к положительно заряженной (trans). Поскольку размер и заряд нуклеотидов различаются, ток через пору изменяется (возникают флуктуации). Эти колебания регистрируются и с помощью распознавания паттернов преобразуются в последовательность ДНК. Основные применения нанопорового секвенатора Polyseq One
Характеристики нанопорового секвенатора Polyseq One
Комплектация нанопорового секвенатора Polyseq One
|
||||||||||||||||||||||||||||||||
|
PY-NSR101
|
|
|
New
|
|
По запросу По запросу По запросу |
|
|
|
||||||||||||||||||||||||
|
PolySeq Hive — это следующий в линейке нанопоровых секвенаторов от компании Polyseq Biotech. В отличии от Polyseq One, оснащен шестью независимыми проточными ячейками. Система сохранила все преимущества предшественника, Polyseq One: простую пробоподготовку, без необходимости докупать реактивы у других производителей и быстрое считывание последовательностей любой длины. Шесть ячеек позволяют проводить шесть параллельных независимых экспериментов, что удобно для лабораторий с большим объёмом исследований. Общий объём информации о последовательности ДНК, полученный с помощью PolySeq Hive, может быть до 360 Гб. Основные применения нанопорого секвенатора PolySeq Hive
Характеристики нанопорого секвенатора PolySeq Hive
Комплектация нанопорого секвенатора PolySeq Hive
|
||||||||||||||||||||||||||||||||
|
Thermo FS
Набор Genexus Barcodes 33-64 AS KIT |
||||||||||||||||||||||||||||||||
|
A40259
|
||||||||||||||||||||||||||||||||
|
Набор содержит один планшет, заполненный 32 одноразовыми бар-кодами, и закрытый прокалываемой фольгой, что исключает риск перекрестного загрязнения между лунками. Бар-коды можно гибко использовать с шагом от 1 до 32, пока не закончатся все бар-коды. При использовании вместе с полосками для библиотек Ion Torrent Genexus 1 и 2AS этот набор обеспечивает гибкую, полностью автоматизированную подготовку и стандартизацию до 32 библиотек Ion AmpliSeq. Применение
Характеристики
Транспортировка и хранение — при комнатной температуре. |
||||||||||||||||||||||||||||||||
|
4405673
|
|
|
|
РУ
Регистрационное удостоверение на медицинское изделие
Росздравнадзора
|
По запросу По запросу По запросу |
|
|
|
||||||||||||||||||||||||
|
Генетический анализатор 3500 — это 8-капиллярный прибор, который может использоваться для широкого спектра задач, включая секвенирование de novo и ресеквенирование (мутационное профилирование), а также микросателлитный анализ, MLPA, LOH, MLST и валидацию или скрининг SNP. Большинство из этих приложений может быть выполнено на одном типе полимера и капиллярной сборке одной длины. Генетические анализаторы серии 3500 были разработаны более 15 лет назад и по настоящее время являются непревзойденными по качеству получаемых данных и удобству использования приборами и являют собой эталон в данной области приборостроения. Серия 3500 использует готовые расходные материалы с предварительно упакованными полимерными пакетами, катодными и анодными буферными контейнерами и легко устанавливаемыми капиллярными сборками. Каждый из этих расходных материалов оснащен RFID-метками, которые позволяют просматривать, отслеживать и сообщать важную информацию о реагентах и расходных материалах, включая данные об использовании, номере партии, номере детали, сроке годности и сроке службы на приборе, в программном обеспечении для сбора данных Data Collection. Программное обеспечение для сбора данных Data Collection отличается удобной навигацией: интуитивно понятный дизайн приборной панели, хорошо заметные кнопки для выполнения общих операций, легко читаемые графические дисплеи для контроля состояния расходных материалов, а также удобный календарь планирования технического обслуживания. Функциональные возможности этого ПО включают упрощенную настройку планшетов и встроенный первичный анализ с контролем качества, что позволяет принимать решения о качестве данных по мере их получения на приборе без необходимости передачи выходных файлов в программные пакеты вторичного анализа. Характеристики генетического анализатора 3500
Комплект поставки: генетический анализатор 3500 (с основным ПО для секвенирования и фрагментного анализа), обучение запуску, управляющий компьютер с установленной программой Data Collection (управление и сбор данных, первичный анализ), кассета для загрузки планшет, инсталляционный комплект для секвенирования и фрагментного анализа. |
||||||||||||||||||||||||||||||||
|
4405633
|
|
|
|
РУ
Регистрационное удостоверение на медицинское изделие
Росздравнадзора
|
По запросу По запросу По запросу |
|
|
|
||||||||||||||||||||||||
|
Генетический анализатор 3500xL — это 24-капиллярный прибор, который может использоваться для широкого спектра задач, включая секвенирование de novo и ресеквенирование (мутационное профилирование), а также микросателлитный анализ, MLPA, LOH, MLST и валидацию или скрининг SNP. Большинство из этих приложений может быть выполнено на одном типе полимера и капиллярной сборке одной длины. Генетические анализаторы серии 3500 были разработаны более 15 лет назад и по настоящее время являются непревзойденными по качеству получаемых данных и удобству использования приборами и являют собой эталон в данной области приборостроения. Серия 3500 использует готовые расходные материалы с предварительно упакованными полимерными пакетами, катодными и анодными буферными контейнерами и легко устанавливаемыми капиллярными сборками. Каждый из этих расходных материалов оснащен RFID-метками, которые позволяют просматривать, отслеживать и сообщать важную информацию о реагентах и расходных материалах, включая данные об использовании, номере партии, номере детали, сроке годности и сроке службы на приборе, в программном обеспечении для сбора данных Data Collection. Программное обеспечение для сбора данных Data Collection отличается удобной навигацией: интуитивно понятный дизайн приборной панели, хорошо заметные кнопки для выполнения общих операций, легко читаемые графические дисплеи для контроля состояния расходных материалов, а также удобный календарь планирования технического обслуживания. Функциональные возможности этого ПО включают упрощенную настройку планшетов и встроенный первичный анализ с контролем качества, что позволяет принимать решения о качестве данных по мере их получения на приборе без необходимости передачи выходных файлов в программные пакеты вторичного анализа. Характеристики генетического анализатора 3500xL
Комплект поставки: генетический анализатор 3500 (с основным ПО для секвенирования и фрагментного анализа), обучение запуску, управляющий компьютер с установленной программой Data Collection (управление и сбор данных, первичный анализ), кассета для загрузки планшет, инсталляционный комплект для секвенирования и фрагментного анализа. |
||||||||||||||||||||||||||||||||
|
HLA 96/7
|
|
96 образцов
|
|
РУ
Регистрационное удостоверение на медицинское изделие
Росздравнадзора
|
По запросу По запросу По запросу |
|
|
|
||||||||||||||||||||||||
|
Наборы Holotype HLA — это современное решение для диагностики и определения человеческих лейкоцитарных антигенов (HLA). Они работают на основе метода секвенирования следующего поколения (NGS) на платформах Illumina и GeneMind. Эти наборы реагентов сочетают в себе высокую точность и удобство использования, позволяют получить информацию о гистосовместимости 5, 7, 11 локусов HLA в зависимости от типа набора. Материал для исследования — образцы геномной ДНК человека. Подготовка образцов
Подготовка библиотеки
Секвенирование
Анализ данных с помощью программного обеспечения HLA TWIN
Технические характеристики Набора Holotype HLA 96/7
|
||||||||||||||||||||||||||||||||
|
HLA 96/5
|
|
96 образцов
|
|
РУ
Регистрационное удостоверение на медицинское изделие
Росздравнадзора
|
По запросу По запросу По запросу |
|
|
|
||||||||||||||||||||||||
|
Наборы Holotype HLA — это современное решение для диагностики и определения человеческих лейкоцитарных антигенов (HLA). Они работают на основе метода секвенирования следующего поколения (NGS) на платформах Illumina и GeneMind. Эти наборы реагентов сочетают в себе высокую точность и удобство использования, позволяют получить информацию о гистосовместимости 5, 7, 11 локусов HLA в зависимости от типа набора. Материал для исследования — образцы геномной ДНК человека. Подготовка образцов
Подготовка библиотеки
Секвенирование
Анализ данных с помощью программного обеспечения HLA TWIN
Технические характеристики Набора Holotype HLA 96/5
|
||||||||||||||||||||||||||||||||
|
HLA 24/7
|
|
24 образца
|
|
РУ
Регистрационное удостоверение на медицинское изделие
Росздравнадзора
|
По запросу По запросу По запросу |
|
|
|
||||||||||||||||||||||||
|
Наборы Holotype HLA — это современное решение для диагностики и определения человеческих лейкоцитарных антигенов (HLA). Они работают на основе метода секвенирования следующего поколения (NGS) на платформах Illumina и GeneMind. Эти наборы реагентов сочетают в себе высокую точность и удобство использования, позволяют получить информацию о гистосовместимости 5, 7, 11 локусов HLA в зависимости от типа набора. Материал для исследования — образцы геномной ДНК человека. Подготовка образцов
Подготовка библиотеки
Секвенирование
Анализ данных с помощью программного обеспечения HLA TWIN
Технические характеристики Набора Holotype HLA 24/7
|
||||||||||||||||||||||||||||||||
|
PY-NSR201
|
|
|
New
|
|
По запросу По запросу По запросу |
|
|
|
||||||||||||||||||||||||
|
Polyseq X2 — высокопроизводительный нанопоровый секвенатор нового поколения от компании Polyseq Biotech, разработанная для решения масштабных геномных задач. Отличается от моделей Polyseq One/Hive технологически усовершенствованной чиповой матрицей, переработанной мембраной и системой стабилизации поровых белков. Адаптированные алгоритмы шумоподавления и улучшенная обработки сигнала обеспечивают высокое качество данных. Максимальная производительность платформы достигает 800 Гб на выходе. Основные применения нанопорого секвенатора Polyseq X2
Характеристики нанопорого секвенатора Polyseq X2
Комплектация нанопорого секвенатора Polyseq X2
|
||||||||||||||||||||||||||||||||
|
Thermo FS
Набор PureLink PCR Purification Kit |
||||||||||||||||||||||||||||||||
|
K310001
|
||||||||||||||||||||||||||||||||
|
Набор обеспечивает более удобный, быстрый и высокопроизводительный процесс очистки по сравнению с аналогами других компаний. После ПЦР-амплификации очистка ПЦР-ампликонов требуется для многих последующих применений (включая гидролиз эндонуклеазами рестрикции, лигирование, секвенирование и мечение ДНК) для того, чтобы предотвратить перенос ферментов (полимеразы, эндонуклеазы рестрикции) и компонентов реакции (dNTPs, праймеры, буферы, соли), которые могут негативно влиять на последующие манипуляции с ДНК. Набор применим только для выделения и очистки линейных (не кольцевых) фрагментов ДНК. Преимущества набора PureLink PCR Purification Kit:
Выделенная ДНК может использоваться для следующих приложений:
Условия хранения и перевозки: при комнатной температуре. |
||||||||||||||||||||||||||||||||
|
Thermo FS
Набор PureLink PCR Micro Kit |
||||||||||||||||||||||||||||||||
|
K310050
|
||||||||||||||||||||||||||||||||
|
Набор идеально подходит для очистки и концентрации ДНК, полученных в ходе низкоэффективных реакций ПЦР или реакций гидролиза рестриктазами. Очищенные фрагменты ДНК и ампликоны пригодны для использования в последующих приложениях, таких как секвенирование ДНК, гидролиз рестриктазами, клонирование, ПЦР. Преимущества набора PureLink PCR Micro Kit:
Выделенная ДНК может использоваться для следующих приложений:
Условия хранения и перевозки: при комнатной температуре. |
||||||||||||||||||||||||||||||||
|
DC203-01
|
|
50 реакций
|
транспортировка +4°C
|
|
7 884, руб.
7 884, руб. RUB
|
|
|
|
||||||||||||||||||||||||
|
Набор предназначен для выделения плазмидной ДНК из 1-5 мл ночной бактериальной культуры. В наборе используется технология спин-колонок и оптимизированные буферные растворы ERB и ERW, что позволяет специфически связывать плазмиду и эффективно удалять эндотоксины, белки и другие примеси. Процесс выделения занимает всего 25 мин. Выделенная ДНК содержит остаток эндотоксина в концентрации менее 0,1 EU/мкг и может быть использована для трансфекций различных клеток и других экспериментов, включая гидролиз эндонуклеазами рестрикции, ПЦР, секвенирование и т.д. Преимущества набора FastPure EndoFree Plasmid Mini
Выделенная ДНК может использоваться для следующих приложений
Условия хранения: комнатная температура. |
||||||||||||||||||||||||||||||||
|
A35892
|
||||||||||||||||||||||||||||||||
|
В наборе используются колонки с усовершенствованными кремнеземными мембранами, которые позволяют выделить плазмидную ДНК пригодную для трансфекции (<1 EU/мкг) за 30 минут. Простой протокол дает возможность получить до 0,4 мг плазмиды, пригодной для стандартной трансфекции и всех методов молекулярной биологии, таких как клонирование и секвенирование.
Преимущества набора PureLink Fast Low-Endotoxin Midi Plasmid Purification Kit:
Выделенная ДНК может использоваться для следующих приложений:
Условия хранения и перевозки: при комнатной температуре. |
||||||||||||||||||||||||||||||||
|
A35895
|
||||||||||||||||||||||||||||||||
|
В наборе для выделения плазмидной ДНК PureLink Fast Low-Endotoxin Maxi Plasmid Purification Kit используются колонки следующего поколения с усовершенствованными кремнеземными мембранами, которые позволяют выделить плазмидную ДНК, пригодную для трансфекции (<1 EU/мкг) примерно за 30 минут. Простой протокол без осаждения дает до 1,5 мг плазмидной ДНК, подходящую для стандартных трансфекций и всех применений в молекулярной биологии, таких как клонирование и секвенирование. Преимущества набора PureLink Expi Endotoxin-Free Mega Plasmid Purification Kit:
Выделенная ДНК может использоваться для следующих приложений:
Условия хранения и перевозки: при комнатной температуре. |
||||||||||||||||||||||||||||||||
|
Oxford Nanopore Technologies
Секвенатор нанопоровый GridION X5 – Capex, 5 x 512 каналов, (аналог арт. PY-NSR101, Polyseq Biotech) |
||||||||||||||||||||||||||||||||
|
GRDCapEx
|
|
|
|
По запросу По запросу По запросу |
|
|
|
|||||||||||||||||||||||||
Характеристки секвенатора нанопорового GridION Mk1GridION X5 – Capex, в отличие от MinION, состоит из пяти проточных ячеек с нанопоровыми каналами и обладает теми же достоинствами, что и MinION – простая пробоподготовка и быстрое секвенирование молекул нуклеиновых кислот любой длины. Пять ячеек позволяют проводить пять параллельных независимых экспериментов, что удобно для лабораторий с большим объёмом исследований. Общий объём информации о последовательности ДНК, полученный с помощью GridION Х5, может быть до 150 Гб (объём встроенного SSD-диска – 4 Тб). В отличие от MinION, в корпус GridION уже встроены все компоненты, необходимые для управления секвенированием и обработки данных:
Комплект поставки: секвенатор GridION X5 – Capex со встроенным ПК. |
||||||||||||||||||||||||||||||||
|
A39513
|
|
|
|
По запросу По запросу По запросу |
|
|
|
|||||||||||||||||||||||||
|
Идентификация видов, присутствующих в образцах пищевых продуктов и кормах, является важным критерием для подтверждения подлинности, проверки происхождения, отслеживаемости сырья и для контроля качества процессов обработки на линиях производства. Рабочий процесс Thermo Scientific NGS Food использует технологию секвенирования следующего поколения Ion Torrent для обеспечения нецелевого подхода к скринингу, позволяющего идентифицировать виды, содержащиеся в пищевых образцах как однокомпонентного, так и многокомпонентного сырья, путем сравнения с базой данных ДНК видов мяса, растений или рыб. Ключевая особенность секвенатора ДНК 2-го поколения Ion GeneStudio S5:
Система Ion GeneStudio S5 использует скорость полупроводникового секвенирования, которая обеспечивает получение высококачественных данных секвенирования за несколько часов и позволяет перейти от библиотеки ДНК к данным всего за 24 часа. Благодаря однодневной установке, простому пользовательскому интерфейсу и картриджам с реагентами система Ion GeneStudio S5 проста и удобна в использовании. Преимущества автоматизированного NGS-скрининга для мультивидовой идентификации:
|
||||||||||||||||||||||||||||||||
|
A35645_компл
|
||||||||||||||||||||||||||||||||
|
Товар временно недоступен. Аналоги по запросу ниже. 4-капиллярный анализатор SeqStudio для секвенирования по Сэнгеру и фрагментного анализа.
Особенности: Универсальный — единый формат расходных материалов (универсальный картридж) и для секвенирования, и фрагментного анализа. Широкий спектр задач — весь спектр приложений капиллярного электрофореза в ваших руках. Компактный — секвенатор размером с амплификатор. Комплексный — компьютер и ПО встроены в прибор. Первичный анализ данных осуществляется в режиме реального времени. Надежный — проверенные временем технологии. Научные, медицинские, криминалистические лаборатории всего мира доверяют данным этих генетических анализаторов. Простой— запуск прибора занимает всего несколько минут. Все необходимое для работы прибора в одном картридже, который хранится непосредственно в приборе. Программное обеспечение:
Комплект поставки: генетический анализатор SeqStudio (с основным ПО), расширенная гарантия (2 года), обучение запуску, ноутбук с установленной программой управления и сбора данных и анализа, стартовый набор для запуска прибора (4-х капиллярный картридж, протектор, катодный буфер, необходимые расходные материалы). Дополнительно: однодневное обучение генетическому анализу. Первые впечатления пользователей нового капиллярного секвенатора SeqStudio (видео), англ. яз.(видео), англ. яз. |
||||||||||||||||||||||||||||||||
|
051.01
|
||||||||||||||||||||||||||||||||
|
Набор для приготовления цепь-специфичных библиотек для секвенирования РНК, включающий все необходимые компоненты. Библиотеки предназначены для следующих задач:
С помощью набора SENSE можно получать библиотеки для секвенирования только на платформе Illumina. Исходным материалом для получения библиотек является РНК после удаления рибосомальной РНК или обогащения поли-А+. Для удаления рибосомальной РНК из образца можно использовать RiboCop rRNA Depletion Kit. Для работы с набором предлагаются два протокола - для интактной РНК и деградированной РНК (например, из FFPЕ-образцов). В состав набора входят все необходимые компоненты для подготовки библиотеки:
|
||||||||||||||||||||||||||||||||
|
050.01
|
||||||||||||||||||||||||||||||||
|
Набор для приготовления цепь-специфичных библиотек для секвенирования РНК, включающий все необходимые компоненты. Библиотеки предназначены для следующих задач:
С помощью набора SENSE можно получать библиотеки для секвенирования только на платформе Illumina. Исходным материалом для получения библиотек является РНК после удаления рибосомальной РНК или обогащения поли-А+. Для удаления рибосомальной РНК из образца можно использовать RiboCop rRNA Depletion Kit. Для работы с набором предлагаются два протокола - для интактной РНК и деградированной РНК (например, из FFPЕ-образцов). В состав набора входят все необходимые компоненты для подготовки библиотеки:
|
||||||||||||||||||||||||||||||||
|
025.03
|
|
1 пробирка
|
|
По запросу По запросу По запросу |
|
|
|
|||||||||||||||||||||||||
|
Набор для приготовления цепь-специфичных библиотек для секвенирования РНК, включающий все необходимые компоненты. Библиотеки предназначены для следующих задач:
С помощью набора SENSE можно получать библиотеки для секвенирования только на платформе Illumina. Исходным материалом для получения библиотек является РНК после удаления рибосомальной РНК или обогащения поли-А+. Для удаления рибосомальной РНК из образца можно использовать RiboCop rRNA Depletion Kit. Для работы с набором предлагаются два протокола - для интактной РНК и деградированной РНК (например, из FFPЕ-образцов). В состав набора входят все необходимые компоненты для подготовки библиотеки:
|
||||||||||||||||||||||||||||||||
|
A41046
|
|
|
|
По запросу По запросу По запросу |
|
|
|
|||||||||||||||||||||||||
|
Товар временно недоступен. Аналоги по запросу ниже. Высокопроизводительная система капиллярного электрофореза.
Программное обеспечение:
|
||||||||||||||||||||||||||||||||
|
A46386_компл
|
|
|
|
По запросу По запросу По запросу |
|
|
|
|||||||||||||||||||||||||
|
Интегрированный секвенатор Ion Torrent Genexus — это высокопроизводительная система секвенирования следующего поколения, которая объединяет подготовку библиотеки, создание шаблонов и секвенирование в однодневный автоматизированный запуск на одном приборе.
|
||||||||||||||||||||||||||||||||
|
RWD Life Science, Китай
Сепаратор клеток на магнитных частицах, со стендом, набором для сепарации клеток |
||||||||||||||||||||||||||||||||
|
CSAK-01
|
|
|
|
81 735, руб.
|
|
|
|
|||||||||||||||||||||||||
|
Продукты для разделения клеток RWD включают в себя наборы для разделения клеток с магнитными микрошариками и колонки для разделения клеток, которые позволяют проводить сепарацию с высокой чистотой и высокой жизнеспособностью за счет простой работы. Наноразмерные магнитные шарики не требуют элюции, а разделенные клетки можно напрямую использовать в последующих экспериментах, таких как проточная цитометрия, культивирование клеток, секвенирование отдельных клеток и т.д. Преимущества набора для магнитной сепарации RWD
Результаты пользователей 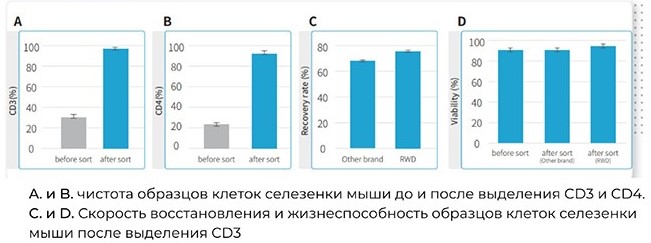 Стандартный протокол магнитной сепарации Стартовый набор для магнитной сепарации включает в себя:
|
||||||||||||||||||||||||||||||||
|
|
||||||||||||||||||||||||||||||||
С помощью личного кабинета Вы сможете:
Сравнение